样品实验方案
简要概述
- 准备样品
- 将75 µL /孔的cAMP标准品或测试样品添加到抗cAMP的96孔板中
- 在室温下孵育5-10分钟
- 加入25 µL /孔的1X HRP-cAMP偶联物
- 在室温下孵育2小时
- 用200 µL /孔的洗涤缓冲液洗涤4次
- 添加100 µL /孔的Amplite Red
- 在室温下孵育15至60分钟
- 检测Ex / Em = 540/590 nm时的荧光增加
溶液配制
储备溶液配制
1. cAMP储备溶液(100 µM):将1 mL测定缓冲液(组分B)添加到cAMP标准品(组分A)的小瓶中。 注意:应将未使用的cAMP储备溶液(100 µM)等分并保存在-20℃下。
2. HRP-cAMP偶联物原液(50X):将55 µL(#36373)或550 µL(#36374)的测定缓冲液(组分B)添加到HRP-cAMP偶联物(组分C)的小瓶中。 注意:未使用的50X HRP-cAMP偶联物原液应分为一次性使用的等分试样,并在-20℃下保存。
3.洗涤液(1倍):将1 mL的10X洗涤溶液(组分D)添加到9 mL蒸馏水中。
4. Amplite Red储备液(200X):将50 µL(#36373)或500 µL(#36374)的DMSO加入到Amplite Red(组分G)的小瓶中。 注意:0.5 µL 200X Amplite Red储备液足以用于一个测定点。 应将未使用的再生原液分装并储存在-20℃。
工作溶液配制
1. HRP-cAMP工作溶液:在使用前,用测定缓冲液(组分B)以1:50稀释,使其具有1X HRP-cAMP偶联物工作溶液。 存放在冰箱或4°C下。 注意:25 µL 1X HRP-cAMP偶联物工作溶液足以用于一个测定点; 准备适当的体积,仅供一次性使用。
2. Amplite Red工作溶液:向10 mL底物缓冲液(组分I)中加入50 µL Amplite Red储备溶液(200X)和11.5 µL 3%H2O2(组分F)注意:Amplite Red工作溶液不稳定,请立即使用。
实验步骤
1.准备样品
1.1根据需要处理细胞:
以下是用Forskolin处理的Hela细胞以96孔板诱导cAMP的示例:吸出细胞生长培养基,在Hanks中加入100 µL /孔100μM Forskolin和20 mM Hepes缓冲液(HHBS),在5%CO2,37°C恒温箱,放置15分钟。孵育后吸出细胞溶液,加入100 µL /孔的细胞裂解缓冲液(组分E),然后在室温下另外孵育10分钟。该细胞裂解液可以直接测定,也可以在测定缓冲液(组分B)中稀释后测定。注意:应单独评估每个细胞系,以确定细胞密度。细胞可以在实验的前或实验当天接种,具体取决于细胞类型和/或受试化合物的作用。
1.2组织样本:
由于组织中环状核苷酸的快速代谢,重要的是在收集后快速冷冻组织(例如,使用液氮)。称重冷冻的组织,并添加10-20 µL / mg细胞裂解缓冲液。在冰箱均质样品。高速旋转5分钟并收集上清液。上清液可以直接测定。
1.3尿液,血浆和培养基样品:
尿液和血浆可以直接在1X裂解缓冲液中以1:200至1:1000的稀释度进行测试。也可以在裂解缓冲液中以1:10至1:200的稀释度测试培养基。注意:RPMI介质可能包含> 350 fmol / µL cAMP。
2.cAMP测定
2.1所有测定孔均按以下顺序准备:A)cAMP标准品,对照或测试样品; B)HRP-cAMP偶联物。
2.2将75 µL /孔的cAMP稀释标准溶液和测试样品添加到抗cAMP Ab包被的96孔板(组分F)的每个孔中。 我们建议对每个标准品和测试样品重复测定。在室温下孵育5至10分钟。
2.3加入25 µL /孔的1X HRP-cAMP偶联物工作溶液。 将板放在摇床上,在室温下孵育2小时。
2.4吸出板内容物,并用200 µL /孔的1X洗涤液洗涤4次。
2.5在每个孔中加入100 µL /孔的Amplite Red工作溶液,并在避光的条件下在室温下孵育10分钟至2小时。
2.6通过使用荧光酶标仪(顶部读取模式),检测Ex / Em = 540/590 nm(截止570 nm)处的荧光增加。
图示
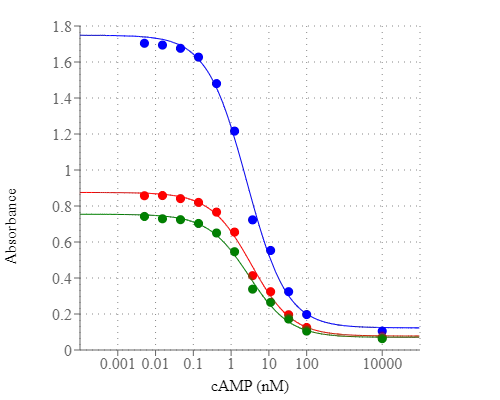
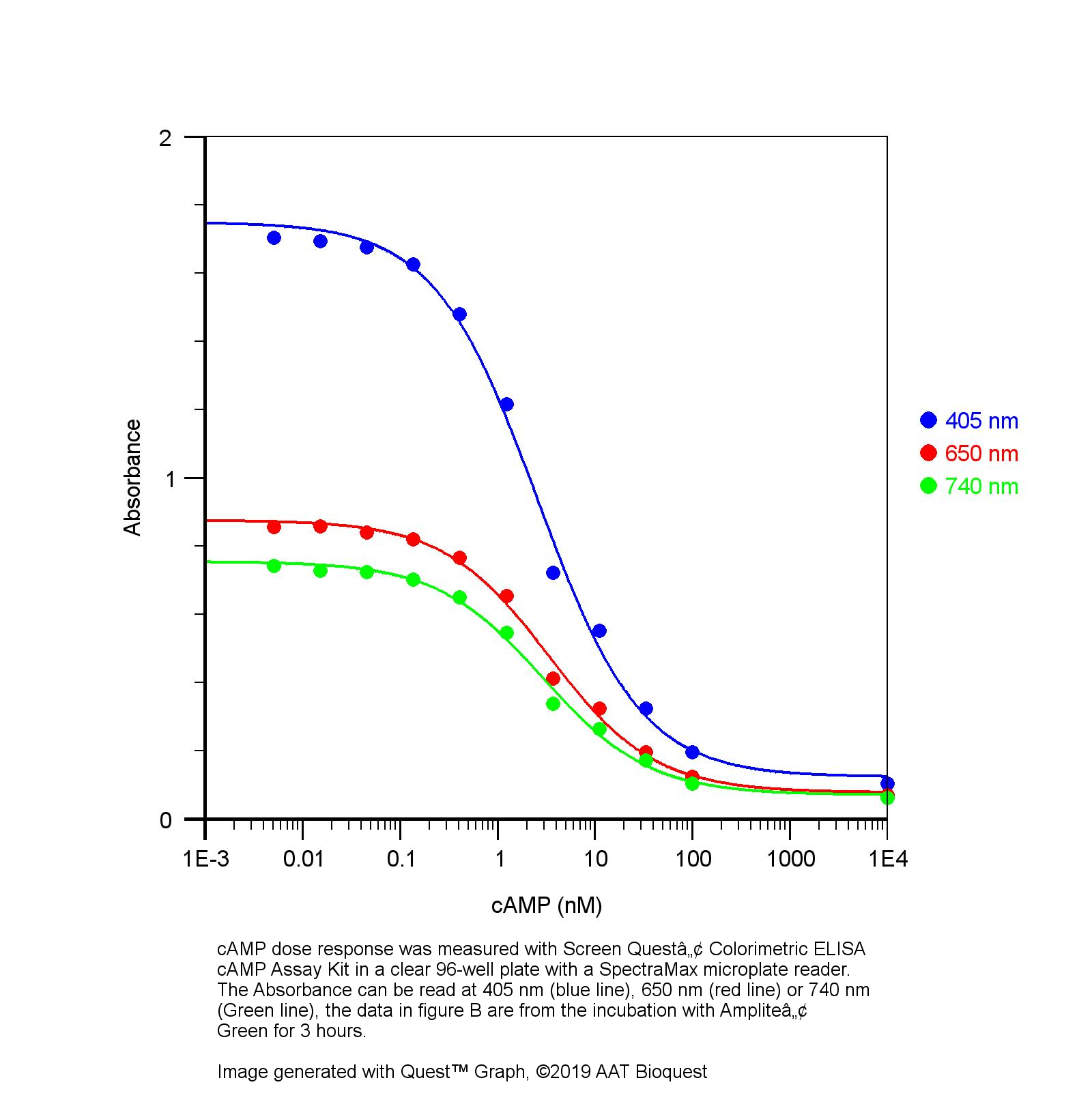
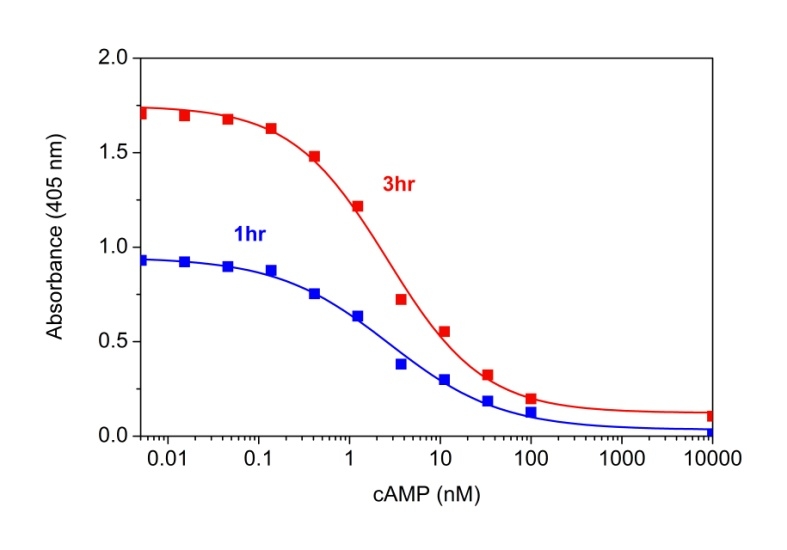
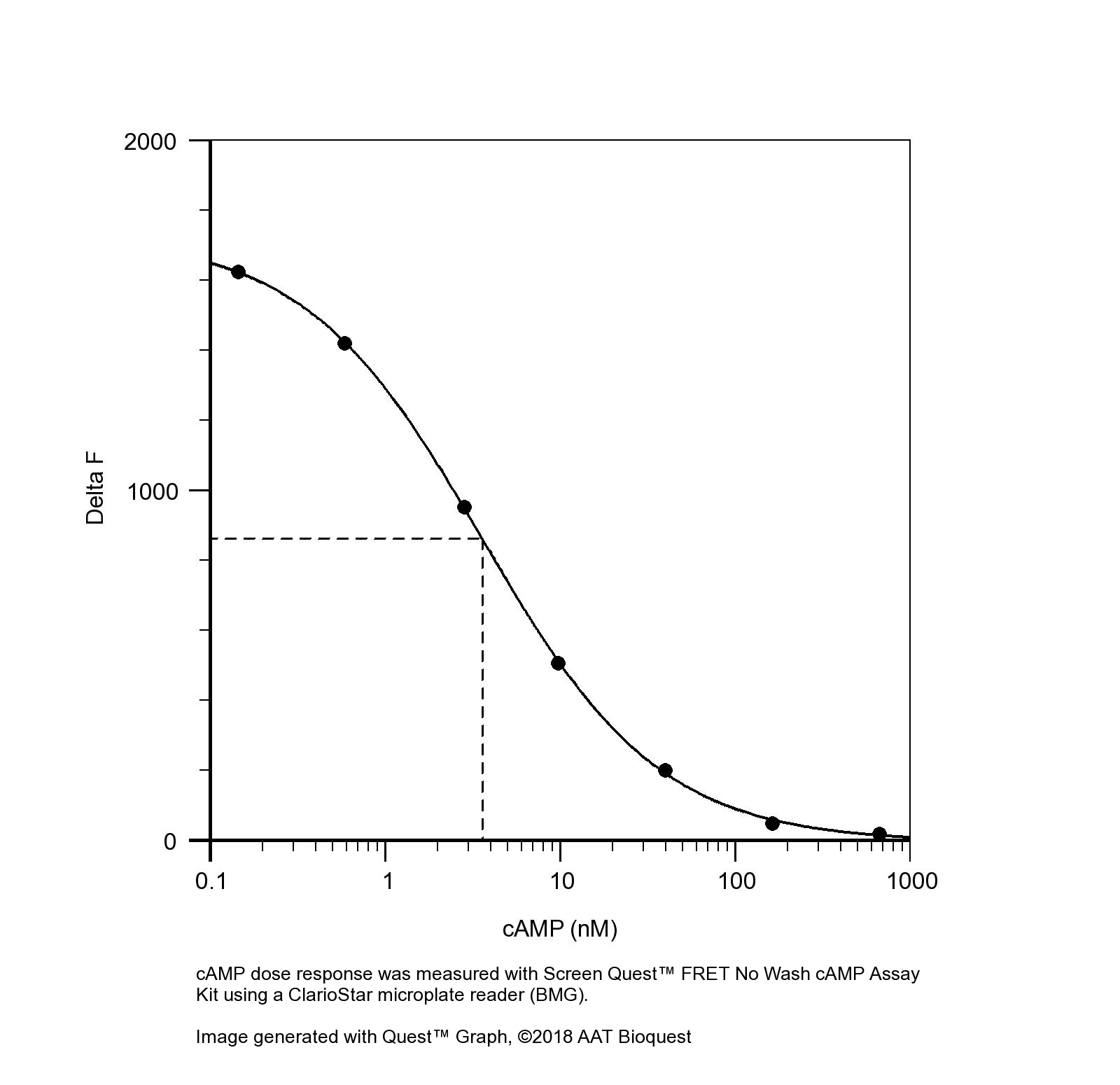
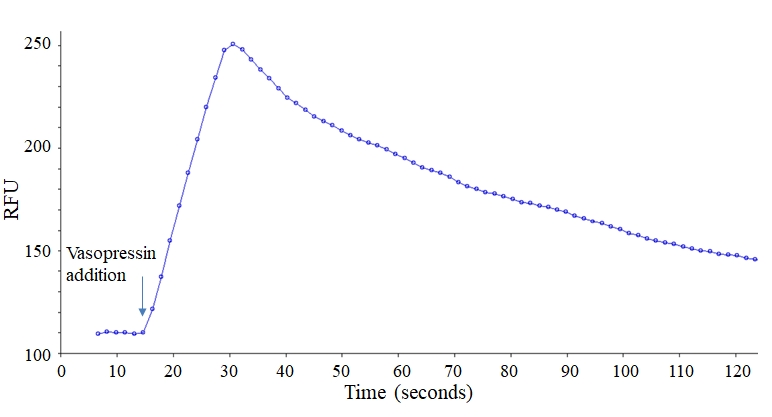
图1.使用Screen Quest 荧光ELISA cAMP测定试剂盒在带有Gemini酶标仪的黑色96孔板上测量cAMP剂量反应。
|
参考文献
A cardiac mitochondrial cAMP signaling pathway regulates calcium accumulation, permeability transition and cell death
Authors: Wang Z, Liu D, Varin A, Nicolas V, Courilleau D, Mateo P, Caubere C, Rouet P, Gomez AM, V and ecasteele G, Fischmeister R, Brenner C.
Journal: Cell Death Dis (2016): e2198
Activation of P2X7 and P2Y11 purinergic receptors inhibits migration and normalizes tumor-derived endothelial cells via cAMP signaling
Authors: Avanzato, D and Genova, T and Pla, A Fiorio and Bernardini, M and Bianco, S and Bussolati, B and Mancardi, D and Giraudo, E and Maione, F and Cassoni, P and others
Journal: Scientific Reports (2016)
Changes in the Arabidopsis thaliana Proteome Implicate cAMP in Biotic and Abiotic Stress Responses and Changes in Energy Metabolism
Authors: Alqurashi M, Gehring C, Marondedze C.
Journal: Int J Mol Sci (2016): 852
Odor-induced cAMP production in Drosophila melanogaster olfactory sensory neurons
Authors: Miazzi F, Hansson BS, Wicher D.
Journal: J Exp Biol (2016): 1798
Role of the cAMP Pathway in Glucose and Lipid Metabolism
Authors: Ravnskjaer K, Madiraju A, Montminy M.
Journal: Handb Exp Pharmacol (2016): 29
The pleiotropic role of exchange protein directly activated by cAMP 1 (EPAC1) in cancer: implications for therapeutic intervention
Authors: Almahariq M, Mei FC, Cheng X.
Journal: Acta Biochim Biophys Sin (Shanghai) (2016): 75
cAMP-Induced Histones H3 Dephosphorylation Is Independent of PKA and MAP Kinase Activations and Correlates With mTOR Inactivation
Authors: Rodriguez P, Rojas J.
Journal: J Cell Biochem (2016): 741
A cAMP Biosensor-Based High-Throughput Screening Assay for Identification of Gs-Coupled GPCR Ligands and Phosphodiesterase Inhibitors
Authors: Vedel L, Brauner-Osborne H, Mathiesen JM.
Journal: J Biomol Screen (2015): 849
Cardiac Hypertrophy Is Inhibited by a Local Pool of cAMP Regulated by Phosphodiesterase 2
Authors: Zoccarato A, Surdo NC, Aronsen JM, Fields LA, Mancuso L, Dodoni G, Stangherlin A, Livie C, Jiang H, Sin YY, Gesellchen F, Terrin A, Baillie GS, Nicklin SA, Graham D, Szabo-Fresnais N, Krall J, V and eput F, Movsesian M, Furlan L, Corsetti V, Hamilton G, Lefkimmiatis K, Sjaastad I, Zaccolo M.
Journal: Circ Res (2015): 707
Cardiac cAMP: production, hydrolysis, modulation and detection
Authors: Boularan C, Gales C.
Journal: Front Pharmacol (2015): 203