上海金畔生物科技有限公司代理AAT Bioquest荧光染料全线产品,欢迎访问AAT Bioquest荧光染料官网了解更多信息。
StrandBrite 绿色RNA定量试剂*200X DMSO溶解 价格 4245
产品规格
产品货号
| Ex (nm) | 509 | Em (nm) | 527 |
| 分子量 | N/A | 溶剂 | DMSO |
| 存储条件 | 在零下15度以下保存, 避免光照 |
StrandBrite 绿色RNA定量试剂是美国AAT Bioquest生产的用于定量RNA的试剂,检测和定量少量RNA对于多种分子生物学程序极为重要,例如在进行Northern blot分析,S1核酸酶测定,RNase保护测定,cDNA文库制备,反向操作之前,测量体外转录RNA的产量和测量RNA浓度。转录PCR和差异展示PCR。测量核酸浓度常用的技术是测定260 nm的吸光度。基于吸收率的方法的主要缺点是蛋白质,游离核苷酸和其他紫外线吸收化合物引起的干扰。使用敏感的荧光核酸染色剂可缓解此干扰问题。StrandBrite RNA定量试剂是一种超灵敏的荧光核酸染料,用于定量溶液中的RNA。StrandBrite RNA定量试剂可通过荧光酶标仪或荧光计检测低至5 ng / mL的RNA。金畔生物是AAT Bioquest的中国代理商,为您提供优质的StrandBrite 绿色RNA定量试剂。
适用仪器
| 荧光酶标仪 | |
| Ex: | 490 nm |
| Em: | 525 nm |
| Cutoff: | 515 nm |
| 推荐孔板: | 纯黑色孔板 |
样品实验方案
溶液配制
工作溶液配制
1.准备StrandBrite 工作溶液:
1.1 准备的水溶液工作溶液StrandBrite 绿色通过在TE浓缩DMSO溶液的200倍稀释液(在DEPC的10mM的Tris-HCl,1mM的EDTA,pH 7.5中经处理的水)。例如,添加50μL StrandBrite 绿色 至10mL TE缓冲液,以制备足够的工作溶液在200至100个测定样品 μ L终体积。通过用箔纸覆盖或将其置于黑暗中来保护工作溶液免受光照。
注意1:我们建议在塑料容器中而不是玻璃中制备此溶液,因为染料可能会吸附到玻璃表面。
注意2:为获得佳结果,应在准备后的几个小时内使用此溶液。
标准液配制:
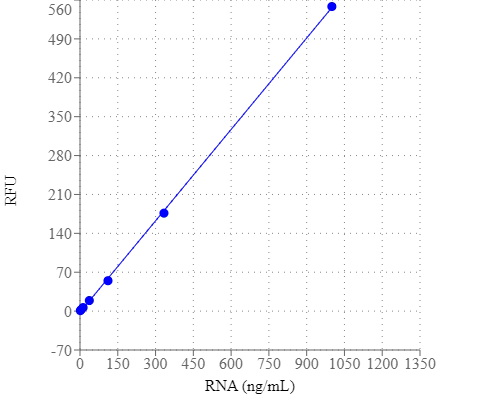
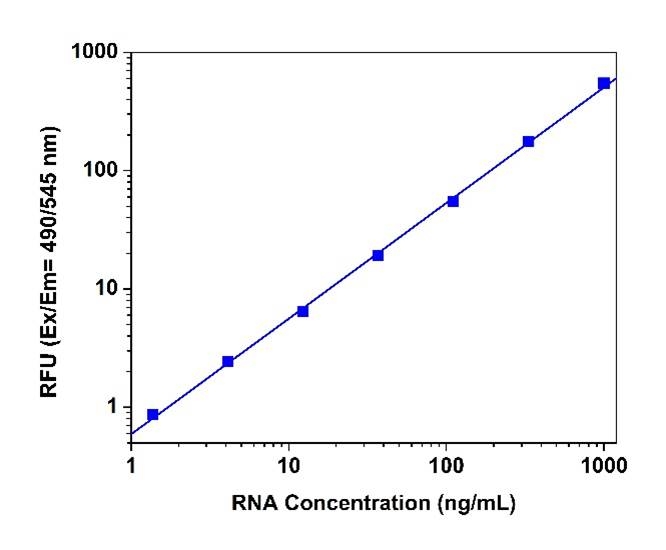
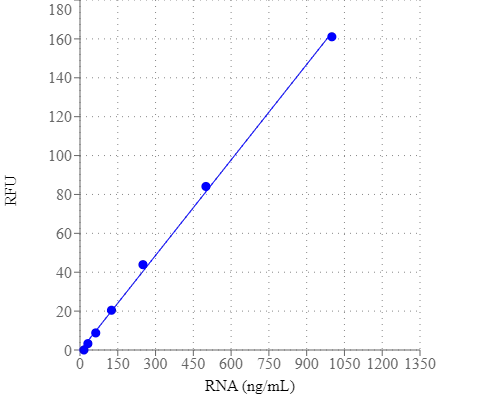
1.准备RNA标准液的系列稀释液(0到1µg / mL ):
1.1准备一个100 μ 克/ mL储备在RNA的溶液DEPC处理过的水。
1.2添加10μL 100 μ 克/毫升RNA储备溶液(来自步骤2.1) ,以490 μ 大号测定缓冲液(成分B)为具有2 微克/ mL的RNA溶液,然后执行1:2系列稀释来获得1000, 500、250、125、62.5、31.3、15.6和0 ng / mL。
1.3如表1和表2所述,将RNA标准品和含RNA 的测试样品添加到96孔黑色固体微孔板中。
表1黑色固体96孔微孔板中RNA标准品和测试样品的布局
| BL | BL | TS | TS |
| RS1 | RS1 | … | … |
| RS2 | RS2 | … | … |
| RS3 | RS3 | ||
| RS4 | RS4 | ||
| RS5 | RS5 | ||
| RS6 | RS6 | ||
| RS7 | RS7 |
注:RS = RNA 标准;BL =空白控制;TS =测试样品
表2每个孔的试剂组成
| RNA标准 | 空白对照 | 测试样品 |
| 连续稀释(100ul) | TL:100ul | 100ul |
注:将从15.6到1000 ng / mL的RNA标准品的系列稀释液一式两份地添加到DS1到DS7的孔中。
运行RNA assay分析:
1.将100μLStrandBrite Green工作溶液(来自步骤2)添加到RNA标准品,空白对照和测试样品(参见步骤3)的每个孔中,以使总RNA测定体积为200μL/孔。
注意:对于384孔板,每孔添加25μL样品和25μLStrandBrite Green工作溶液。
2.在避光的条件下,将反应在室温下孵育5至10分钟。
3.用分光荧光计在Ex / Em = 490/525 nm (在515 nm 处截止)监测荧光的增加。
注意:为使光漂白效应小化,请对所有样品保持恒定的荧光测量时间。
4.用空白孔(仅含测定缓冲液)中的荧光作为对照,并从具有RNA标准液或测试样品的比色皿中减去。RNA样品的浓度根据RNA标准曲线测定。
参考文献
Inhibitors of Streptococcus pneumoniae surface endonuclease EndA discovered by high-throughput screening using a PicoGreen fluorescence assay
Authors: Peterson EJ, Kireev D, Moon AF, Midon M, Janzen WP, Pingoud A, Pedersen LC, Singleton SF.
Journal: J Biomol Screen (2013): 247
Validation of a PicoGreen-based DNA quantification integrated in an RNA extraction method for two-dimensional and three-dimensional cell cultures
Authors: Chen Y, Sonnaert M, Roberts SJ, Luyten FP, Schrooten J.
Journal: Tissue Eng Part C Methods (2012): 444
Characterization of PicoGreen interaction with dsDNA and the origin of its fluorescence enhancement upon binding
Authors: Dragan AI, Casas-Finet JR, Bishop ES, Strouse RJ, Schenerman MA, Geddes CD.
Journal: Biophys J (2010): 3010
Comparison of SYBR Green I-, PicoGreen-, and [3H]-hypoxanthine-based assays for in vitro antimalarial screening of plants from Nigerian ethnomedicine
Authors: Abiodun OO, Gbotosho GO, Ajaiyeoba EO, Happi CT, Hofer S, Wittlin S, Sowunmi A, Brun R, Oduola AM.
Journal: Parasitol Res (2010): 933
Metal-enhanced PicoGreen fluorescence: application to fast and ultra-sensitive pg/ml DNA quantitation
Authors: Dragan AI, Bishop ES, Casas-Finet JR, Strouse RJ, Schenerman MA, Geddes CD.
Journal: J Immunol Methods (2010): 95
Quantification of dsDNA using the Hitachi F-7000 Fluorescence Spectrophotometer and PicoGreen dye
Authors: Moreno LA, Cox KL.
Journal: J Vis Exp. (2010)
Development and characterization of a novel host cell DNA assay using ultra-sensitive fluorescent nucleic acid stain “PicoGreen”
Authors: Ikeda Y, Iwakiri S, Yoshimori T.
Journal: J Pharm Biomed Anal (2009): 997
Enhanced DNA dynamics due to cationic reagents, topological states of dsDNA and high mobility group box 1 as probed by PicoGreen
Authors: Noothi SK, Kombrabail M, Kundu TK, Krishnamoorthy G, Rao BJ.
Journal: FEBS J (2009): 541
Factors affecting quantification of total DNA by UV spectroscopy and PicoGreen fluorescence
Authors: Holden MJ, Haynes RJ, Rabb SA, Satija N, Yang K, Blasic JR, Jr.
Journal: J Agric Food Chem (2009): 7221
Label-free DNA sequence detection with enhanced sensitivity and selectivity using cationic conjugated polymers and PicoGreen
Authors: Ren X, Xu QH.
Journal: Langmuir (2009): 43